| 独立行政法人 理化学研究所 神戸研究所 発生・再生科学総合研究センター |
| 2008年9月24日 |
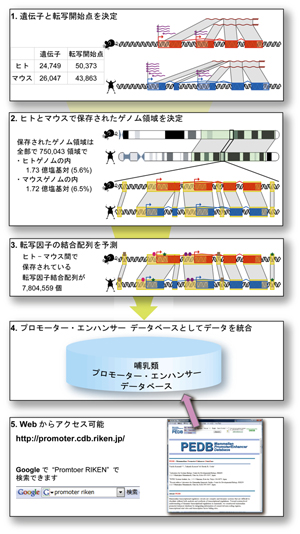
我々の睡眠周期など、日・季節・年などを通して変化する生物のリズムは体内時計と呼ばれるシステムにより調節されており、様々な生理機能に関与している。この体内時計というシステムからの制御によって、遺伝子は周期的な発現を繰り返している。例えば概日リズムでは、「朝」「昼」「夜」、それぞれの時間に遺伝子の発現を制御するDNA配列(制御配列)が存在し、これらの制御配列がスイッチの役割を果たすことで、さまざまな遺伝子が24時間周期の発現を繰り返している。しかし、この「朝」「昼」「夜」の制御配列がゲノム上にどれだけ存在し、どのような遺伝子の発現を制御しているのかは不明の部分が多かった。 今回、理研CDBシステムバイオロジー研究チーム(上田泰己チームリーダー)の熊木勇一客員研究員(本務:株式会社インテックシステム研究所)、鵜飼-蓼沼磨貴テクニカルスタッフらは、マウス及びヒトゲノムに存在する遺伝子の発現制御領域(プロモーター領域とエンハンサー領域)のデータベースを構築し、「朝」「昼」「夜」の遺伝子発現のための制御配列を網羅、同定することに成功した。更にコンピューターを用いて「朝」「昼」「夜」の制御配列を人工的に設計、生物学的実験により検証し、実際に細胞内で制御配列として働くことを実証した。この研究は株式会社インテックシステム研究所、近畿大学の重吉康史教授、ペンシルバニア大学のジョン・ホゲネシュ(John B. Hogenesch)准教授らのチームとの共同で行われ、研究成果は米国科学アカデミー紀要(Proceedings of the National Academy of Sciences of the United States of America: PNAS)に9月24日付けでオンライン公開された。 遺伝子が発現する量や時期は、ゲノム上でその遺伝子近傍にあるプロモーター領域とエンハンサー領域と呼ばれる特別なゲノム領域によって制御されている。熊木客員研究員らは、ヒトとマウスのゲノム配列を比較し、進化の過程を通じて種間で保存されているゲノム領域を解析することなどにより、遺伝子の発現制御領域であるプロモーター領域とエンハンサー領域を、約25,000もの遺伝子について網羅し、データベース化した。概要は図1の通りである。データベースはhttp://promoter.cdb.riken.jp/にて非営利目的の研究者に対して無料で公開されている。また、今回の発表した論文ではヒトとマウスの比較についてのみ記載されているが、公開中のデータベースではヒトとラットの比較データについても閲覧可能である。
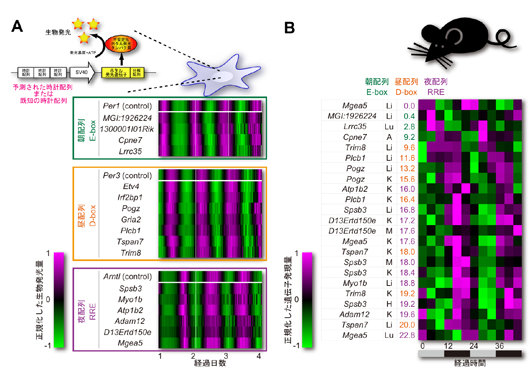
熊木客員研究員らはこのデータベースを用いることで、体内時計の制御下にある「朝」「昼」「夜」の制御配列をゲノムから探索した。朝配列(E-box)、昼配列(D-box)、夜配列(RRE)、それぞれのコンセンサス配列(共通している配列)は既知であり、その配列はゲノム上に多数存在するが、その中で実際に制御配列として機能する配列は一部のみである。そこで、「隠れマルコフモデル」という統計学的手法を用いて、これまでに機能することがわかっている既知の制御配列である朝配列(E-box)、昼配列(D-box)、夜配列(RRE)の特徴を抽出したモデルを構築した。このモデルとデータベースを組み合わせて、ヒトとマウスに共通して存在している制御配列を検索する方法を用いることで、機能していることがより期待される1,108の朝配列、2,314の昼配列、3,288の夜配列をゲノム上に同定した。これらの配列は http://promoter.cdb.riken.jp/circadian.htmlに公開されている。
次に熊木客員研究員らは、「朝」「昼」「夜」の制御配列を探索するのに用いたモデルを応用することで、朝配列・昼配列・夜配列として機能することが期待される人工的な制御配列を設計した。また、逆に機能しないことが期待される人工的な制御配列についても設計した。これらの人工配列について生物学的実験で検証を行ったところ、「機能することが期待される人工配列」は実際の細胞内で「機能する人工配列」であり、人工配列が実際の細胞内でも「朝」「昼」「夜」配列それぞれの周期的な発現を制御し、遺伝子発現のスイッチとなり得ることを証明した。一方、「機能しないことが期待される人工配列」は実際の細胞内で「機能しない人工配列」であり、はっきりした周期性を持った発現が示されなかった。この違いは如何にしておこるのか更に解析を進めたところ、昼配列と夜配列の「機能する人工配列」には、転写活性化因子及び転写抑制因子が強く結合し、逆に「機能しない人工配列」では転写活性化因子及び転写抑制因子が弱く結合することが分かった。ところが朝配列では、転写抑制因子が「機能する人工配列」と「機能しない人工配列」のどちらにも同定度に強く結合したが、転写活性化因子は「機能する人工配列」よりも「機能しない人工配列」により強く結合することが分かった。さらにコンピューターシミュレーションによる解析を行った結果、転写活性化因子と転写抑制因子が配列に結合する際の、結合の強さのバランスが重要であることが示唆された。 今回の研究によって、哺乳類 プロモーター・エンハンサー データベースを構築し用いることで、体内時計による「朝」「昼」「夜」の遺伝子発現制御がゲノムレベルで明らかになってきたといえる。また、このデータベースは今回の解析以外にも様々な生命現象に関わる遺伝子発現制御機構の解析に非常に有効であると考えられる。そして、今回の研究で用いられたコンピューターによる解析と生物学的実験を組み合わせた解析方法は、生命現象を研究する新たな解析法として有効な方法となることが期待される。
|
||||||||||||||||||||||