| 独立行政法人 理化学研究所 神戸研究所 発生・再生科学総合研究センター |
| 2008年10月29日 |
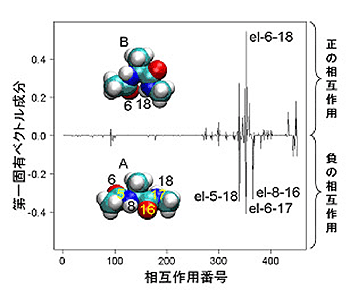
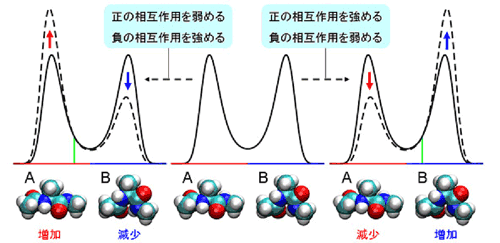
タンパク質やDNAといった生体内の分子は、その構造を変化させることで様々な機能を発揮している。このような構造変化を解析するために、個々の原子間の相互作用を計算して分子の安定性や動きを予測する「分子動力学シミュレーション」が幅広く用いられている。しかし、原子の位置(原子座標)を用いた従来の解析手法「主成分分析(PCA)」では、複数の安定な構造の間を遷移するような大きな構造変化を解析することが困難であった。 今回、理研CDBの小山洋平研修生(システムバイオロジー研究チーム、上田泰己チームリーダー)らは、従来の原子座標を用いた主成分分析(PCA)を一般化し、原子間の相互作用のエネルギーを用いた主成分分析(PEPCA: potential energy PCA)を開発した。この方法を用いることで分子の大きな構造変化を解析・予測できるようになった。この研究はPhysical Review E誌に10月7日付けで発表された。 従来の原子座標を用いたPCAは、特定の安定な構造のまわりでの「揺らぎ」を解析するのに適しているが、複数の安定構造を遷移するような大きな構造の変化を解析するのには適していなかった。分子の構造変化だけを抽出するためには、あらかじめ分子全体の運動(並進運動と回転運動)を取り除く必要があるが、大きな構造変化を伴う場合、回転運動を取り除くのが難しいためだ。また、摂動(温度変化や薬剤などの結合による環境の変化)によって引き起こされる分子の構造変化を予測する理論も知られているが、同様の問題を抱えていた。 そこで、小山らはまず、従来の原子座標を用いたPCAを一般化し、任意の物理量(原子座標の関数)に対するPCAが、分子の最も大きな構造変化を引き起こす摂動を探す手法として解釈できることを明らかにした。次に、PCAにおける上述の問題を解決するために、物理量として原子座標そのものではなく、原子間の相互作用のエネルギー(ポテンシャルエネルギー)を用いた主成分分析「ポテンシャルエネルギー主成分分析(PEPCA: potential energy PCA)」を開発した。ポテンシャルエネルギーは静電相互作用などからなり物理的な意味が明確である。また、原子間の相対的な位置だけに依存するため、その値は分子全体の運動には影響されない。すなわち、あらかじめ分子全体の運動を取り除く必要が無く、従来の問題を回避することができる。 小山らはPEPCAの有効性を実証するために、2つの安定状態を遷移するアラニンジペプチドをモデル分子として解析を行なった。その結果、最も大きい構造変化を引き起こす相互作用の組合せ(図1)が2つの安定状態の比率を変化させることが分かり(図2)、それぞれの安定状態に重要な正および負の相互作用が同定された(図1)。同様に、2番目に大きい構造変化を引き起こす相互作用の組合せが2つの安定状態とその間の遷移状態の比率を変化させることが分かった。このように、PEPCAを適用することにより、2つの安定状態とその間の遷移状態、およびそれぞれの構造に重要な原子間相互作用が体系的に同定できることがわかった。
上田チームリーダーは、「近年、大規模並列計算機の高速化や専用計算機の開発により分子動力学シミュレーションで生体分子の機能に関わるような大きな構造変化をシミュレートできるようになりつつあります。今後、このような大きな構造変化に対して今回のPEPCAを適用することで、分子機能に重要な原子間相互作用を同定することが可能になると考えられます。このような相互作用をターゲットにした薬剤の開発、つまり、構造変化の予測に基づいて分子機能を制御するような薬剤の開発が将来可能になると期待されます」と展望を語った。
|