| 独立行政法人 理化学研究所 神戸研究所 発生・再生科学総合研究センター |
2013年3月28日 |
生物の基本単位は細胞である。そのため生命現象を検証するためには、細胞集団を対象とした解析に加え、個々の細胞がどのように振る舞い、それがどのように全体を形成しているのかを知る必要がある。特に集団内の細胞が相互作用を通して多様化していく発生過程を理解するためには、それぞれの細胞でどのような遺伝子が発現しているのかをその空間配置と併せて知る事が重要となってくる。現在一般的にFISH (fluorescence in situ hybridization)という方法が用いられ個々のRNA分子の配置を可視化できるようになったが、一度に解析できる遺伝子数が限られるなど限界があった。 理研CDBのLars Martin Jakt 研究員(幹細胞研究グループ、西川伸一グループディレクター)らは、 一般的なFISH法に改良を加え、高解像度・高感度顕微鏡を組み合わせる事により、固定試料において細胞内の一つ一つのRNA分子を6種類以上同時に可視化する手法を開発した。またこの方法を用いることで、中胚葉から血管内皮が分化する際の個々の細胞の遺伝子発現の移り変わりが明らかになった。この研究結果はDevelopment 誌の1月号に掲載された。
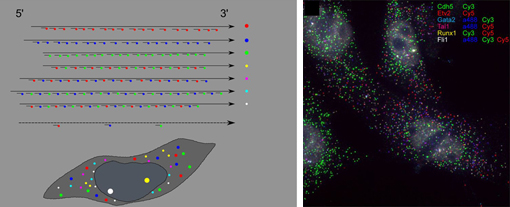
FISH法は蛍光ラベルを付加したRNAプローブを細胞内のmRNAに結合させることで、細胞内で発現している遺伝子の種類と量を解析する手法である。これを改良して一つの遺伝子に対して多数の短いプローブを用いると蛍光を増幅できるうえに比較的低コストであるという利点があったが、まだ3つの遺伝子までしか同時に解析できないという欠点があった。そこで研究グループはさらに改良を加え、3種類の蛍光を組み合わせて1つの分子を標識することで、同定できる遺伝子数を3種類から6種類に増やした。この方法を利用すると1つの分子に対するプローブの種類が多いので擬陽性の影響が少なく、検出精度をあげることも出来る。また研究グループはこの解析に適した高解像度・高感度の顕微鏡を用いることで、実際に6種の同時発現解析が高精度に行えることを確認した。 彼らはこの方法を用いて中胚葉から血管内皮への分化過程における、各細胞の遺伝子発現プロフィールの移り変わりの可視化を試みた。これまでの研究より、血管内皮へと分化する中胚葉の細胞はEtv2を発現しており、内皮特異的な遺伝子群の発現を誘導することが示唆されていた。また内皮の細胞が続いて分化する造血幹細胞に必要なTal1, Fli1, Gata2 といった遺伝子を直接誘導することも知られていた。しかしこれまでの細胞集団を対象とした解析では、個々の細胞の分化のタイミングに違いがあるため各遺伝子がどのような順序でEtv2に誘導されるのかを知るのは困難であった。 そこでES細胞に分化誘導をかけて内皮に分化させる過程において、それに関わる6つの遺伝子(Tal1, Runx1, Fli1, Gata2, Cdh5, Etv2)に対してcandyFISHを行い、個々の細胞内のRNA分子を可視化した像を得た。ある一点の時間における細胞ごとの遺伝子発現の割合と濃度を円グラフにして2次元に表すと細胞群をいくつかの集団に分けることができた。これらをもとに解釈すると初めにEtv2 の発現がおこりそれに続いてTal1, Fli1, Gata2 が同時に誘導され、一時的に発現が高まるがその後低下し、それと共にCdh5が発現してくるといった、一連の発現順序の詳細が明らかになった。これより細胞間の遺伝子発現のバラつきは分化への発現変化様式の差によるものではなく、分化の過程に入るタイミングがそれぞれ違うためであることが示唆された。 以上のことより、一度の解析で細胞内の多数のRNA発現を可視化することの有用性が証明され、この手法を他に応用することにより様々な解析に使用できることが想像される。この手法は使用可能な蛍光の数や使う顕微鏡の解像度に依存するが、現在において可能な設備では既に10個かそれ以上の遺伝子について同時解析が可能と考えられる。また今後顕微鏡の精度の向上などと共にさらにその数は増えていき、組織の深部の解析も可能になっていくであろうことが予想される。
|
||||||||||||||
 |
| Copyright (C) CENTER FOR DEVELOPMENTAL BIOLOGY All rights reserved. |