| 独立行政法人理化学研究所 発生・再生科学総合研究センター |
2013年4月26日 |
睡眠は多くの動物にとって必要不可欠な生理現象の一つである。睡眠状態は主にノンレム睡眠(NREM: Non-Rapid eye movement)、レム睡眠(REM: Rapid eye movement)、覚醒の3状態に分類され、これらは脳波と筋電図の波形によって定義される。睡眠や睡眠障害のメカニズムを理解するためには、睡眠状態を詳しく解析する必要があるが、これまで一つの大きな障壁があった。睡眠状態の判定に人が介入するため、主観が入ってしまう上に、解析の大規模化が難しかったのだ。 理研CDBの砂川玄志郎研究生(システムバイオロジー研究プロジェクト、上田泰己プロジェクトリーダー)らは、マウスをモデルにした研究で、睡眠状態を全自動解析できるFASTER法を開発し、より客観性の高い睡眠判定を可能にするとともに、大規模な睡眠解析への道を開いた。この研究は徳島大学大学院、大阪バイオサイエンス研究所、日本大学薬学部などとの共同で行われ、Genes to Cells 誌のオンライン版に4月29日付けで発表される予定。
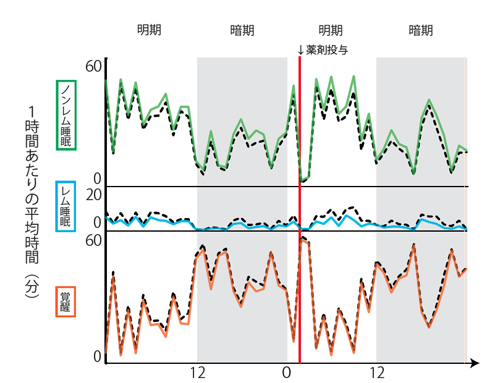
睡眠時に得られる脳波や筋電図の特徴は個体ごとに異なり、マウスでは特徴が現れる周波数域も系統によって異なることが知られている。そのため、通常の睡眠解析では、一定時間(例えば24時間)脳波と筋電図のデータをとった上で、判定者がデータ全体を見ながら個体ごとに睡眠3状態の判定基準を設定する必要がある。判定基準の設定に人が介入することで、個体間の差を考慮したりノイズなどを除外できる一方、判定者の主観が入ったり誤判定が生じたりする。また、人が介入すると多くの個体を対象にした大規模な解析が難しくなり、解析に時間も要する。これまでにも、コンピュータを用いた半自動睡眠判定法は開発されているが、肝心の判定基準の設定は人が実験毎に行う必要があった。 今回砂川らは、睡眠状態を判定する際の判定基準を予め固定することで睡眠判定を完全自動化し、判定者の主観が入る余地を排除したFASTER法(Fully Automated Sleep sTaging method via EEG/EMG Recordings)を開発した 。一方で、判定基準を固定すると、個体間の差を考慮した解析が行えない、ノイズなどの不確定データを除外できないという問題が生じる。FASTER法では幾つかの工夫を加えることで判定する睡眠データを予め最適化し、これらの問題を解決している。まず脳波の解析において、通常使われている周波数帯域だけでなく、記録している全周波数帯域を解析対象にすることで、個体間で異なる周波数帯域を広くカバーした。これにより解析データは膨大になるが、主成分分析を行うことで特徴的な情報を効率よく抽出するようにした。また、単位時間(マウスの場合は通常8秒)毎の睡眠データを判定するのではなく、ノンパラメトリック密度推定クラスタリングという方法で予め特徴の似た睡眠データをグループ化し、グループの特徴を判定することによって3状態の境界領域にある睡眠データも判別しやすくした。これらの工夫により、FASTER法では予め判定基準を固定しながらも、個体特有のデータ分布や未知のデータ分布も解析可能になった。 彼らはFASTER法の精度の検証も行った。まず、野生型マウスの睡眠状態を従来法で判定した結果を正解モデルに設定し、FASTER法で用いる各パラメーターの校正を行った。次に、薬剤で睡眠状態を変容させたマウスや体内時計遺伝子を欠損させたマウスを用い、従来法とFASTER法で睡眠判定を行った。その結果、両者の解析結果は90%以上一致するという結果が得られ、FASTER法の高い精度が示された。また、従来法では解析に1〜2時間要するのに対し、完全自動化したFASTER法では一般的なノートパソコンを用いて約10分で解析できた。 FASTER法が実用化されれば、大規模かつより定量的な睡眠解析が可能になり、動物を用いた薬剤スクリーニングなどに活用できる可能性がある。ただし、「FASTER法だけで大規模解析ができるわけではありません。マウスへの電極付加など人手のかかる物理的な作業もあります。そのような物理作業を簡略化する方法も現在研究しています」と砂川氏は語る。さらに、「FASTER法では大量のデータから全自動で的確な睡眠判定ができます。ヒトの場合はより多くの睡眠データを扱いますので、今回のFASTER法はマウスだけでなく、ヒトの睡眠解析にも応用できると期待しています」と話す。
|
||||||||||||||
 |
| Copyright (C) CENTER FOR DEVELOPMENTAL BIOLOGY All rights reserved. |